
- DAZ.online
- DAZ / AZ
- DAZ 41/2005
- Epigenetik, ...
Molekularbiologie
Epigenetik, Biomodulatoren und Krankheiten
Was ist Epigenetik?
Der Begriff "Epigenetik" wurde 1953 von dem englischen Biologen Conrad H. Waddington geprägt. Damit wollte er eine Erklärung für zahlreiche Befunde geben, denen zufolge in verschiedenen Stadien der Entwicklung unterschiedliche Muster der Genexpression auftreten. Dass dem Organismus zu verschiedenen Zeitpunkten seiner Entwicklung spezifische genetische Informationen selektiv zur Verfügung stehen, führte Waddington auf "eine komplexe Interaktion von Genen mit ihrer Umwelt" zurück. Sollte es doch eine Vererbung erworbener Eigenschaften geben, wie sie der französische Naturforscher Lamarck vor 150 Jahren postuliert hatte?
In den letzten 20 Jahren ist der molekularbiologische Hintergrund des zellulären Geschehens sehr viel deutlicher geworden, aber in seiner Gesamtheit noch nicht vollständig verstanden, besonders was die pathogenen Aspekte betrifft. Auch der Einfluss bestimmter Umweltfaktoren auf Veränderungen der genetischen Information ist noch weitgehend unbekannt.
Das Humane Epigenomprojekt will die vererbbaren Veränderungen in den Expressionsmustern von Genen erfassen und damit auch die Basis für die Entwicklung neuartiger diagnostischer Methoden und Therapieansätze, z. B. bei Krebserkrankungen oder Diabetes, ermöglichen. Epigenetik ist heute als "Modifikation einer DNA-Funktion ohne Veränderung der DNA-Sequenz" definiert [1].
Stumme und aktive Gene
In somatischen Zellen liegt die DNA in Form von Nukleosomen vor. Dabei ist die fädige DNA um die Histone, einen Komplex spezifischer basischer Proteine, gewickelt. Mehrere Nukleosomen werden zum Chromatin vernetzt. Bereits auf dieser Ebene wird die Genaktivität reguliert, denn es gibt einerseits
- das Heterochromatin, das so stark verschnürt ist, dass die genetischen Informationen nicht abgelesen werden können, und andererseits
- das Euchromatin, das weniger stark verschnürt, sodass die Gene bei Bedarf wieder angeschaltet werden können.
Das Euchromatin umfasst stumme (engl. silenced) und aktive Gene; sie werden über epigenetische Modifikationen reguliert, die entweder die Histone oder die DNA selbst betreffen [2].
Epigenetische Modifikationen
Eine der epigenetischen Modifikationen erfolgt über die Methylierung des Cytosins in Cytidin-Guanosin-Dinukleotiden (CpG; p bedeutet Phosphodiester), die sich gehäuft in bestimmten DNA-Abschnitten (CpG-Inseln) finden. An diesem Vorgang sind mehrere DNA-Methyltransferasen (DNA-MT oder DNMT) beteiligt: Er wird durch DNMT3a und -3b katalysiert und bei weiteren Zellteilungen durch DNMT1 konserviert. Die Methylierung eines DNA-Abschnitts blockiert dessen Transkription (engl. "Silencing"; Abb. 1).
Eine Reihe anderer epigenetischer Modifikationen setzt bei den Histonen an. Die N-terminalen Enden der Histonproteine ragen aus dem nukleosomalen Kern heraus und dienen als Targets für zahlreiche regulatorische Eingriffe, z. B. die Acetylierung von Lysin, die Phosphorylierung von Serin und Threonin oder die Verknüpfung von Lysinresten mit Ubichinon. Die Gesamtpalette dieser Regulationen wird als Histon-Code bezeichnet.
Die Kombination von DNA-Methylierung und Histon-Deacetylierung in spezifischen DNA-Abschnitten ist ein wirkungsvoller Mechanismus für das dauerhafte Abschalten von Genen (Abb. 1). Es stellt sich natürlich die Frage, welchen Sinn dies hat. Bei den abgeschalteten Genen handelt es sich großenteils um "junk"-DNA, repetitive Sequenzen, die sich im Laufe der Evolution in unser Genom eingeschlichen haben, und zwar vorwiegend durch virale Infektionen.
Man schätzt, dass etwa 45% des menschlichen Genoms aus viralen Retrotransposons und endogenen Retroviren bestehen, die durch das Genom "wandern". Das "Silencing" ist damit ein Schutzmechanismus gegen die unerwünschte Expression dieser Gene. Auch für bestimmte Entwicklungsabläufe im Organismus sind epigenetische Regulationsvorgänge unerlässlich [3].
DNA-Methylierungsmuster und Krebs
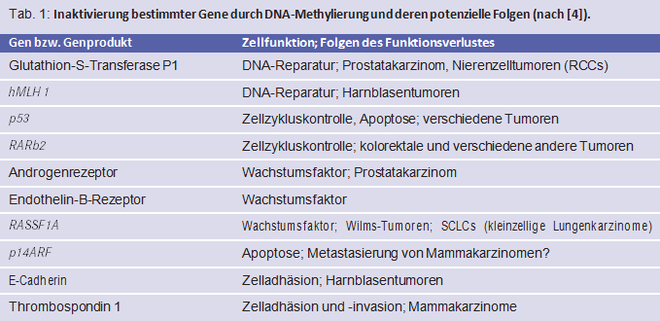
Epigenetische Modifikationen spielen eine Rolle bei der Karzinogenese [4 - 6]. Einen ersten Hinweis darauf gab der Befund, dass bei Neoplasien eine generelle Hypomethylierung des Genoms besteht [7]. Dann wurde nachgewiesen, dass die Hypomethylierung regional ausgeprägt ist und einige an der Tumorgenese und -progression beteiligte Gene betrifft. Andererseits zeigte sich, dass bei einigen Tumoren spezifische Gene hypermethyliert sind. Ein Methylierungsmuster von nur drei bis vier Genen reicht bereits aus, um einen Tumor mit einer Spezifität von 70 bis 90% zu definieren [4].
Mit kolorektalen Tumoren ist häufig das DNA-Methylierungsmuster "CpG island methylator phenotype" (CIMP) assoziiert [8]. Hier sind die Promotoren mehrerer Tumorsuppressorgene methyliert und inaktiviert (der Promotor ist ein DNA-Abschnitt, der die Transkription des ihm benachbarten Gens kontrolliert). Letztlich sind die Zusammenhänge beim CIMP aber noch komplexer:
Es gibt hier zwei Wege der DNA-Methylierung,
- eine altersabhängige, die auch in gesundem Gewebe auftritt, sowie
- eine pathogene, die bisher nur in den kolorektalen Tumoren nachgewiesen wurde.
Darüber hinaus besteht vermutlich auch ein Zusammenhang zwischen CIMP und der Inaktivierung von Genen, die die Metastasierung oder die Angiogenese von Tumorgewebe unterdrücken. Tatsächlich werden multiple Tumorsuppressorgene wie p16 und DNA-Reparaturgene wie hMLH1 gleichzeitig inaktiviert. Einige Beispiele für die Rolle, die die DNA-Methylierung und die daraus folgende Inaktivierung bestimmter Gene bei der Tumorbildung spielt, seien genannt:
Die Inaktivierung
- des Tumorsuppressorgens p53 hemmt die Apoptose von Krebszellen und tritt bei verschiedenen Tumoren auf.
- des Gens BRCA1 induziert Doppelstrangbrüche der DNA. In der Folge können Mamma- und Ovarialkarzinome entstehen. Defekte des BRCA1 werden für etwa 90% der Fälle von erblichem Brustkrebs verantwortlich gemacht.
- des O6-Methyl-Guanin-Methlytransferase-Gens (MGMT), das vor der Zytotoxizität alkylierender Substanzen schützt, kann zu Kolonkarzinomen führen.
- eines DNA-Reparaturgens, das Fehler bei der DNA-Replikation korrigiert, führt zu Mutationen in den Mikrosatellitensequenzen, die ein bestimmtes Kolonkarzinom verursachen können.
Es gibt zahlreiche weitere Beispiele für eine krebspathogene DNA-Methylierung (Tab. 1) [4]. Da jede Krebserkrankung ein spezifisches DNA-Methylierungsmuster besitzt, lässt sie sich durch den Nachweis der tumorspezifischen DNA im Serum erkennen; dabei kommt die methylierungsspezifische Polymerase-Kettenreaktion (MSP) zum Einsatz [4]. Solche Gentests könnten die Früherkennung von Krebs verbessern.
Epigenetik auch bei anderen Erkrankungen?
"Der langsame Fortschritt bei der Aufklärung der genetischen Grundlagen komplexer Erkrankungen könnte sich auf die Grenzen konventioneller Untersuchungsstrategien, die sich auf die Suche nach entsprechenden DNA-Mutationen oder -Polymorphismen beschränken, zurückführen lassen", argumentiert etwas provokativ der kanadische Neurogenetiker A. Petronis [9]. Er geht davon aus, dass eine Reihe von Erkrankungen - zumindest teilweise - auf eine epigenetische Fehlregulation von Genen zurückzuführen ist. So könnten die unterschiedlichen Erkrankungsphasen der Multiplen Sklerose mit epigenetischen Änderungen zusammenhängen; auch für bestimmte psychische Erkrankungen könnte dies zutreffen.
Unter der launigen Überschrift "Spur der Currywurst" berichtete die Frankfurter Allgemeine Sonntagszeitung am 17.11.2002, dass "jede Wurst in sogenannten epigenetischen Mustern auf der DNA verewigt" wird. Richtig ist, dass sich bestimmte Umwelteinflüsse - so auch eine ungesunde Ernährung - auf die DNA-Methylierungsmuster auswirken und möglicherweise Stoffwechselerkrankungen auslösen. Deshalb steht auch der Diabetes Typ 2 auf der Liste der Erkrankungen, die man künftig mit Hilfe von Gentests diagnostizieren zu können hofft.
Gestützt wird diese Hoffnung durch Tierversuche amerikanischer Wissenschaftler: Mäuse, die aufgrund eines - auch von der Katzenzüchtung bekannten - mutierten Agouti-Gens übergewichtig waren und ein gelbes Fell hatten, brachten z. T. schlanke, braune Junge zur Welt, wenn sie während der Schwangerschaft ein mit verschiedenen Nährstoffen angereichertes Futter erhalten hatten. In diesen Fällen hatten die Nährstoffe DNA-Methyltransferasen aktiviert, die das Agouti-Gen abgeschaltet haben [10].
Eine retrospektive schwedische Studie kam zu dem Ergebnis, dass die Ernährungsgewohnheiten männlicher Vorfahren einen Einfluss auf kardiovaskuläre Erkrankungen und Diabetes haben [11]. Ferner scheint ein geringes Geburtsgewicht, das auf Stress während der fetalen Entwicklung zurückzuführen ist, mit dem Auftreten von Diabetes Typ 2 im Erwachsenenalter zu korrelieren.
Epigenetische Therapie mit molekularen Schaltern?
Während genetische Veränderungen in der Regel irreversibel sind, lassen sich epigenetisch inaktivierte Gene durch bestimmte Moleküle potenziell wieder "anschalten"; solche spezifischen Biomodulatoren oder Chemosensitizer heißen deshalb auch "molekulare Schalter". Beispielsweise können demethylierende Substanzen wie 5-Azacytidin und 5-Aza-2'-deoxycytidin eine durch DNA-Methylierung erworbene Resistenz gegen Chemotherapeutika wieder aufheben. Sie können aber auch zu einer verstärkten Expression von Onko- und Protoonkogenen führen. Im konkreten Fall sind die Wechselwirkung zwischen Biomodulatoren, Onkogenen, Tumorsuppressorgenen und DNA-Reparaturgenen jeweils genau zu untersuchen [4].
Auch Histon-Deacetylase-Inhibitoren wie Trichostatin A könnten bestimmte epigenetisch inaktivierte Gene gezielt wieder "anschalten". Es ist auch nicht auszuschließen, dass sich die Wirksamkeit einiger bereits bekannter chemopräventiver Substanzen mit einer epigenetischen Regulation erklären lässt [13].
Da für unterschiedliche Tumoren bislang recht widersprüchliche Befunde vorliegen und auch die zugrundeliegenden Faktoren der Tumorentstehung und -progression nicht vollständig verstanden sind, werden epigenetische Therapieansätze wohl noch einige Zeit auf sich warten lassen. Es ist aber unbestritten, dass epigenetische Veränderungen zu den frühesten Auffälligkeiten während der Karzinogenese zählen und dass sie damit ein hervorragendes Target für den diagnostischen Nachweis und potenziell auch für chemopräventative Maßnahmen darstellen.
Anschrift des Verfassers:
Dr. Hans-Peter Hanssen,
Technologie-Beratung,
Sillemstraße 83, 20257
Hamburg
E-Mail: hans-peter.hanssen@hamburg.de
Literatur
[1] Jones, P.A., Takai, D., The role of DNA methylation in mam- malian epigenetics. Science 293 (2001) 1068 –1070.
[2]Jaenisch, R., Bird, A., Epigenetic regulation of gene expres- sion: how the genome integrates intrinsic and environmental signals. Nature Genetics 33 Suppl. (2003) 245 –254.
[3]Li, P., Chromatin modification and epigenetic reprogramming in mammalian development. Nature Rev. Genetics 3 (2002) 662 –673.
[4] Arnold, C., Usadel, H., Blum, H.E., Epigenetik: Bedeutung für Tumorentstehung und Klinik. Dtsch. Med. Wochenschr. 127 (2002) 1701 –1703.
[5] Allegrucci, C., Thurston, A., Lucas, E., Young, L., Epigenetics and the germline. Reproduction 129 (2005) 137 –149.
[6] Lund, A.H., v. Lohuizen, M., Epigenetics and cancer. Genes & Development 18 (2004) 2315 –2335.
[7]Chen, R.Z., Pettersson, U., Beard, C., Jackson-Grusby, L., Jaenisch, R., DNA hypomethylation leads to elevated muta- tion rates. Nature 395 (6697) (1998) 89 –93.
[8] Toyota, M., Ahuja, N., Ohe-Toyota, M., Herman, J.G., Baylin, S.P., Issa, J.-P.J., CpG island methylator phenotype in colo- rectal cancer. Proc. Natl. Acad. Sci. USA 96 (15) (1999)
8681 –8686.
[9] Petronis, A., Human morbid genetics revisited: relevance of epigenetics. Trends Genetics 17 (2001) 142 –146.
[10] Gießen, H., Das Genom über dem Genom. Pharm. Ztg. 148
(2003) 4420.
[11] Kaati, G., Bygren, L.O., Edvinsson, S., Cardiovascular and diabetes mortality determined by nutrition during parents’ and grandparents’ slow growth period. Eur. J. Hum. Genetics 10 (2002) 682 –688.
[12] www.epigenome.org.
[13] Kopelovich, L., Crowell, J.A., Fay, J.R., The epigenome as a target for cancer chemoprevention. J. Natl. Cancer Inst. 95 (2003) 1747 –1757.
Das Humane Epigenom-projekt (HEP) Mehrere Forschungsinstitute in Europa gründeten 1999 das Human Epigenome Consortium (HEC). Nach einer erfolgreichen Pilotstudie, die im 5. Rahmenprogramm für Forschung und technologische Entwicklung der EU gefördert worden war, begann im Oktober 2003 das Humane Epigenomprojekt (HEP), das für eine Laufzeit von fünf Jahren konzipiert ist.
Aufgabe des HEP ist es, Abschnitte im Humangenom zu identifizieren und zu charakterisieren, deren Cytosinbasen durch DNA-Methylierung modifiziert sind. Die gewonnenen Daten werden sukzessive veröffentlicht [12].




















0 Kommentare
Das Kommentieren ist aktuell nicht möglich.