
- DAZ.online
- DAZ / AZ
- DAZ 3/2023
- Next-Generation-Vakzine ...
COVID-19
Next-Generation-Vakzine gegen COVID-19
Was den neuen Impfstoffkandidaten BNT162b4 auszeichnet
Das Genom der SARS-CoV-2-Varianten gleicht einer Spielwiese, auf der man sich austoben kann. Nachdem mit BNT162b einmal das Prinzip erfolgreich etabliert wurde, mit einer modifizierten mRNA-Sequenz das Immunsystem gegen das Virus zu stimulieren, können die verschiedenen Optimierungsstrategien angegangen werden. Mittlerweile verliert man schon fast den Überblick über die verschiedenen Abkürzungen, die alle mit BNT162b beginnen (s. Tabelle). Leider lässt sich nicht immer eindeutig und leicht nachvollziehen, was genau hinter dem „verbesserten Präfusions-Spike-Protein“ in BNT162b5 oder der „T-Zell-Antigen-mRNA“ in BNT162b4 steckt. Nachdem der Markt der mRNA-Impfstoffe heiß umkämpft ist, ist es natürlich eine Frage der Patentierbarkeit, was in welcher Form wem preisgegeben werden kann. Während BNT162b5 mit der Variation in der Präfusionsform des Spike-Proteins wahrscheinlich nur eine Abwandlung des bereits zugelassenen Impfstoffes ist, wird mit BNT162b4 ein neuer Ansatz verfolgt: Zum einen geht es um Nicht-Spike-Proteine und zum anderen kommen T-Zell-Epitope ins Spiel.
Kürzel | Sequenz / Besonderheit |
|---|---|
BNT162b | Sequenz für das Spike-Protein des SARS-CoV-2-Isolats Wuhan-Hu-1 |
BNT162b1 | Sequenz für die Rezeptor-Bindedomäne des Spike-Proteins |
BNT162b2 | Sequenz für das komplette Spike-Protein mit einer Mutation, die das Protein in der Präfusionskonformation fixiert |
BNT162b3 | Sequenz für die transmembranäre Rezeptor-Bindedomäne des Spike-Proteins |
BNT162b4 | Sequenz für Nicht-Spike-Proteine von SARS-CoV-2 mit T-Zell-Epitopen |
BNT162b5 | Bivalent mit Sequenz für das Spike-Protein des Wildtyp-Virus und der Omikron-Variante BA.2 in einer verbesserten Präfusionskonformation |
Das Besondere an BNT162b4
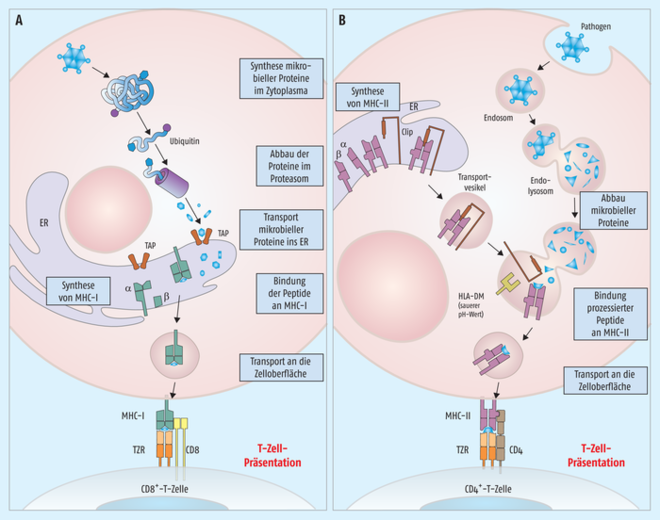
Auch wenn darüber ebenfalls nichts Konkretes publiziert wurde, außer den üblichen Pressemitteilungen, lässt sich doch eine interessante Überlegung nachvollziehen. Die bisherigen mRNA-Impfstoffe fokussieren sich alle auf das Spike-Protein. Nach Verabreichung der Vakzine wird das Spike-Protein anhand der Information aus der mRNA synthetisiert und sezerniert. Das virale Protein kann anschließend von antigenpräsentierenden Zellen phagozytiert und auf MHC(major histocompatibility complex)-II-Molekülen präsentiert werden. In der Folge kommt es schließlich zur Bildung von Antikörpern gegen das Spike-Protein – idealerweise auch von neutralisierenden Antikörpern, die die Infektion weiterer Körperzellen verhindern können. Allerdings hat sich inzwischen gezeigt, dass durch Mutationsereignisse neue Virusvarianten entstehen, die sich gerade auch im Spike-Protein unterscheiden und dadurch den neutralisierenden Antikörpern entkommen können. Eine zweite Schiene der induzierten Immunantwort ist die Aktivierung zytotoxischer T-Zellen. Durch die Bildung des Spike-Proteins in unseren Körperzellen werden durch das Proteasom erzeugte Fragmente des Proteins über MHC-I präsentiert. Das aktiviert wiederum zytotoxische CD8+-T-Zellen, die infizierten Körperzellen zu bekämpfen (s. Abb. 1).
Abb. 1: Unterschiedliche Präsentation eines Peptid-Antigens über MHC-I und MHC-II-Moleküle. A) Während über MHC-I Teile von intrazellulär synthetisierten Proteinen präsentiert werden, tragen B) MHC-II-Moleküle Fragmente von phagozytierten Pathogenen in ihrer Bindungstasche.
Das Protein-Repertoire von SARS-CoV-2
Natürlich werden die entsprechenden Immunreaktionen auch gegen andere virale Proteine ausgelöst, sobald die jeweiligen kodierenden mRNAs als Impfstoff verabreicht werden. In der Datenbank UniProtKB sind für SARS-CoV-2 insgesamt 17 Proteine registriert. Vier davon sind Strukturproteine: das Spike(S)-, das Envelope(E)-, das Nucleo(N)- und das Membran(M)-Protein. Alle anderen kodierten Proteine werden als Nicht-Strukturproteine (NSP) bezeichnet, darunter auch die virale RNA-abhängige RNA-Polymerase und zwei Proteasen. Allerdings ist davon auszugehen, dass mehr als diese 17 Proteine bei der Virusreplikation entstehen: Zum einen konnten noch einige zusätzliche offene Leserahmen auf dem RNA-Genom identifiziert werden, zum anderen zerschneiden die viralen Proteasen die großen Polyproteine in kleinere Einheiten. Inzwischen sind 16 kleinere Nicht-Strukturproteine bekannt und neun sogenannte akzessorische Proteine, die von verschiedenen offenen Leserahmen am 3‘-Ende des Virus-Genoms kodiert werden. Von etlichen dieser Proteine weiß man, dass sie in zelluläre Mechanismen der Virus-Abwehr eingreifen und für die Ausbreitung der Viren im Körper wichtig sind. Sequenzvergleiche zwischen Nicht-Strukturproteinen verschiedener Virus-Varianten zeigen, dass diese NSP (bisher) stark konserviert sind. Impfstoffe, die gerade die konservierten Proteine adressieren, könnten also eine Immunantwort induzieren, die vor ganz unterschiedlichen Virusvarianten schützt.
Zu beachten ist allerdings, dass die bei einer Verimpfung von NSP-mRNAs gebildeten Antikörper keinen Schutz bieten, weil sie nicht in der Lage sind, die Virus-Partikel zu neutralisieren: Die Nicht-Strukturproteine sind nicht auf der Oberfläche der Viren präsent und nicht an der Bindung an die Wirtszelle beteiligt. Stattdessen zielt eine derartige Impfstrategie darauf ab, zytotoxische CD8+-T-Zellen zu mobilisieren, die wiederum die infizierten Zellen abtöten. Damit dies möglichst effizient passiert, kommt der zweite Aspekt bei BNT162b4 zum Tragen: die T-Zell-Epitope.
T-Zell-Epitope
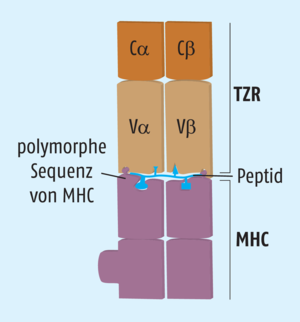
Zytotoxische CD8+-T-Zellen erkennen ein Antigen über ihren T-Zell-Rezeptor (TZR). Jeder TZR besteht aus zwei Ketten, die jeweils eine variable und eine konstante Region besitzen. Ähnlich wie bei B-Zellrezeptoren/Antikörpern entsteht bei der Entwicklung der T-Zellen durch zufällige, kombinatorische Rearrangements der kodierenden Gensegmente eine Vielzahl verschiedener T-Zell-Rezeptoren. Schätzungen zufolge gibt es im menschlichen Körper ungefähr 1013 unterschiedliche T-Zell-Klone, die jeweils über ein spezifisches Antigen aktiviert werden können. Allerdings muss dieses Antigen von anderen Zellen über MHC-Moleküle präsentiert werden. Für die Antigen-Erkennung durch T-Zell-Rezeptoren auf zytotoxischen CD8+-T-Zellen sind die MHC-I-Moleküle nötig, die sich auf allen kernhaltigen Körperzellen befinden. Im Menschen werden die MHC-Moleküle auch als humane Leukozytenantigene (HLA) bezeichnet, sind zwischen den Individuen sehr unterschiedlich und deshalb für die Gewebeverträglichkeit bei Transplantationen verantwortlich. MHC-I sind generell so aufgebaut, dass in einer „Grube“ Peptide mit einer Länge von acht bis 14 Aminosäuren Platz finden. Inzwischen kennt man für etliche MHC-I-Varianten diejenigen Aminosäuren, die an bestimmten Positionen in der Peptid-Sequenz vorkommen sollten, damit die Antigene bevorzugt präsentiert werden (s. Abb. 2). In der Folge entsteht wiederum eine Oberfläche des Peptids, die in einen T-Zell-Rezeptor passen und ihn aktivieren muss. Die Auswahl des präsentierten Antigens findet also über das jeweilige MHC-I-Molekül statt. Allerdings ist dadurch noch nicht gewährleistet, dass das Antigen auch ausreichend immunogen ist. Auch die Interaktion des Peptid-MHC-I-Komplexes mit dem T-Zell-Rezeptor wurde bereits genauer analysiert. Daraus ließen sich Algorithmen wie EpiMatrix ableiten, mit deren Hilfe die Genome von Pathogenen auf passende T-Zell-Epitope hin durchforstet werden können. Beachtet werden muss dabei aber, dass diese Sequenzen keine Homologien zu humanen Proteinen aufweisen, ansonsten würde es zu einer Immuntoleranz gegenüber dem verabreichten Impfstoff kommen.
MHC und TZR – Eine lebenswichtige Beziehung
Im Zusammenhang mit den Impfungen gegen COVID-19 wurde unser immunologisches Grundwissen gefordert: Oft genug war die Rede davon, dass wir neben den neutralisierenden Antikörpern auch die spezifischen T-Zellen brauchen, um wirklich vor einer schweren Erkrankung geschützt zu sein. Bei den T-Zellen brauchen wir zum einen die T-Helferzellen, die an dem Oberflächenmolekül CD4 zu erkennen sind (CD4+-T-Zellen) und die B-Zellen dabei unterstützen, hochaffine Antikörper gegen das Impfantigen zu machen. Zum anderen werden zytotoxische T-Zellen gebraucht, erkennbar am Oberflächenmolekül CD8 (CD8+-T-Zellen), die dafür sorgen, dass infizierte Körperzellen abgetötet werden. Damit die T-Zellen wissen, gegen welches Antigen sie vorgehen sollen, tragen sie einen passenden Rezeptor auf ihrer Oberfläche, an den das Antigen bindet. Natürlich „wissen“ unsere T-Zellen nicht von Anfang an, welche Antigene ihnen im Laufe unseres Lebens begegnen werden, um dafür einen passenden T-Zell-Rezeptor vorzuhalten. Stattdessen entstehen während der T-Zellentwicklung ganz viele T-Zellklone, die sich jeweils in ihren T-Zell-Rezeptoren unterscheiden. Man schätzt, dass durch komplexe Gen-Rearrangements ca. 1013 unterschiedliche T-Zell-Rezeptoren gebildet werden, sodass im Prinzip jedes mögliche Antigen, das uns irgendwann einmal attackiert, erkannt und gebunden werden kann.
T-Zell-Rezeptoren können aber nicht jedes „daherkommende“ Antigen einfach so binden. Vielmehr brauchen sie das Antigen in der passenden Größe und in einem passenden Rahmen, den die sogenannten MHC-Moleküle vorgeben. In allen Wirbeltieren finden sich zwei verschiedene Major Histocompatibility Complex (MHC) Klassen: MHC-I und MHC-II. Im Menschen bezeichnet man die MHC-Moleküle auch als humane Leukozyten-Antigene (HLA), nachdem man entdeckt hatte, dass diese Moleküle für Gewebeunverträglichkeiten relevant sind. Die MHC-Moleküle sind innerhalb der Bevölkerung enorm divers. Schätzungen zufolge gibt es ca. 10.500 verschiedene humane MHC-I- und 3500 MHC-II-Moleküle. Jedes Individuum exprimiert jedoch nur jeweils die Moleküle, die es von der Mutter bzw. vom Vater geerbt hat, also nur jeweils zwei MHC-I- und MHC-II-Varianten. Während sich die MHC-I-Proteine auf der Oberfläche jeder unserer kernhaltigen Zellen befinden und eine wichtige Rolle bei der Erkennung des eigenen, intakten Gewebes spielen, exprimieren nur dendritische Zellen, Makrophagen und B-Lymphozyten MHC-II-Moleküle. Jedes MHC-Molekül hat eine Bindungstasche, in die es kurze Peptidsequenzen einlagern kann (s. Abb. 2). Je nach Aussehen dieser Tasche erklärt es sich, dass Peptide mit bestimmten Aminosäuren an Schlüsselpositionen bevorzugt „präsentiert“ werden. Inzwischen kennt man die „Vorlieben“ einiger der polymorphen MHC-Moleküle und kann Vorhersagen treffen, wie gut ein bestimmtes Protein als Antigen verarbeitet wird.
Wie kommt es zur Beladung der MHC-Moleküle? Hier gibt es wieder den Unterschied zwischen MHC-I und MHC-II (s. Abb. 1). MHC-I-Moleküle bekommen Peptide, die aus dem zellulären Proteinbiosyntheseweg stammen und beispielsweise wegen einer Fehlfaltung im zellulären „Mülleimer“, dem Proteasom, zerschnitten wurden. Beladen mit dem Peptid findet der Komplex anschließend den Weg an die Zelloberfläche. Ist beispielsweise eine Körperzelle mit einem Virus infiziert und bildet entsprechend Virus-Proteine, finden sich virale Peptide als Antigene in den MHC-I-Molekülen dieser Zelle. Demgegenüber werden MHC-II-Moleküle mit Peptiden beladen, die von Lysosomen der phagozytierenden Immunzellen stammen. Hat beispielsweise eine dendritische Zelle ein Pathogen „gefressen“, gelangt das Bakterium oder Virus über das Endosom in das Endolysosom, wo es von den verschiedenen verdauenden Enzymen zerkleinert wird. Bruchstücke des Pathogens werden dann in die MHC-II-Moleküle eingelagert und gelangen so an die Zelloberfläche.
Jetzt fehlt nur noch die Beziehung zwischen TZR und MHC. Durch die entsprechenden Co-Rezeptoren des T-Zell-Rezeptors auf den CD4+-T-Zellen bzw. CD8+-T-Zellen passen die T-Zell-Rezeptoren auf den CD4+-T-Zellen nur mit den MHC-II-Molekülen zusammen, während die T-Zell-Rezeptoren auf CD8+-T-Zellen nur mit MHC-I-Molekülen interagieren können. Dadurch wird sichergestellt, dass die zytotoxischen CD8+-T-Zellen effizient beispielsweise virusinfizierte Körperzellen erkennen und sicher eliminieren. Ein absolut cleveres Prinzip!
Fazit
BNT162b4 ist ein sehr spannender Ansatz, um sehr gezielt mit SARS-CoV-2 infizierte Zellen zu eliminieren und dadurch die weitere Ausbreitung der Viren zu verhindern. Eine Kombination dieser Vakzine mit dem Standard-mRNA-Impfstoff gegen das Spike-Protein könnte also einen umfassend(er)en Schutz vor COVID-19 bieten. Natürlich muss sich erst noch zeigen, ob die T-Zell-Epitope wirklich eine robuste Immunantwort der CD8+-T-Zellen induzieren. |
Literatur
De Groot AS, Moise L, Terry F et al. Better Epitope Discovery, Precision Immune Engineering, and Accelerated Vaccine Design Using Immunoinformatics Tools. Front Immunol 2020;11:442
Joglekar AV, Li G. T cell antigen discovery. Nature Methods 2021;18:873-880
Low ZY, Zabidi NZ, Yip AJW et al. SARS-CoV-2 Non-Structural Proteins and Their Roles in Host Immune Evasion. Viruses 2022;14:1991
Pfizer and Biontech Advance Next-Generation COVID-19 Vaccine Strategy with Study Start of Candidate Aimed at Enhancing Breadth of T cell Responses and Duration of Protection. Pressemitteilung der Pfizer Inc. vom 16. November 2022, www.pfizer.com/news/announcements/pfizer-and-biontech-advance-next-generation-covid-19-vaccine-strategy-study
Schaap-Johansen A-L, Vujovic M, Borch A et al. T Cell Epitope Prediction and Its Application to Immunotherapy. Front Immunol 2021;12:712488























0 Kommentare
Das Kommentieren ist aktuell nicht möglich.